Dask vs Pandas : Fight !
Chaque semaine, Deepki accompagne de nouveaux clients dans la transition énergétique et gère un volume de plus en plus important de données. Dans ce contexte de croissance il m’a paru judicieux d’explorer les Dask DataFrames afin de prévoir une alternative à Pandas. Dans cet article, je vous présenterai une série de tests comparatifs entre Dask et Pandas dans l’objectif de mettre en évidence les différences en temps d’éxecution entre les deux librairies.

Dask
Dask est une librairie conçue pour le traitement de sets de données volumineux et le parallélisme en Python.
Elle est composées de deux parties : Dynamic task scheduling et Big Data collection.
Cette dernière partie gère des arrays, dataframes et lists qui héritent d’interfaces communes tels que Numpy, Pandas ou
des itérateurs Python.
Cet article porte ponctuellement sur les Dask DataFrames. Ces derniers sont des grands dataframes composés de plusieurs dataframes Pandas. Ainsi, un traitement sur un dataframe Dask se traduit par des tâches en parallèle sur plusieurs dataframes et par conséquent des temps de traitement plus rapides.
Geonames
Pour les différents tests, j’ai utilisé la base de données géographique Geonames. Elle contient plus de 12 millions de “points d’intérêt” dans le monde (1.5GB) et elle en libre accès au format txt.
Round 1 : Lecture de données
Le premier test porte sur la lecture de la base de données et sa conversion en DataFrame. Dans cet objectif, j’ai écrit deux fonctions équivalentes : la première avec Dask et la deuxième avec Pandas.
import dask.dataframe as dd
import pandas as pd
def test_geonames_dask_txt():
df = dd.read_csv('allCountries.txt', sep='\t',
dtype={9: 'object', 10: 'object', 11: 'object',
12: 'object', 13: 'object' },
header=None)
return df
def test_geonames_pandas_txt():
df = pd.read_table('allCountries.txt', sep='\t',
dtype={9: 'object', 10: 'object', 11: 'object',
12: 'object', 13: 'object'},
header=None)
return df
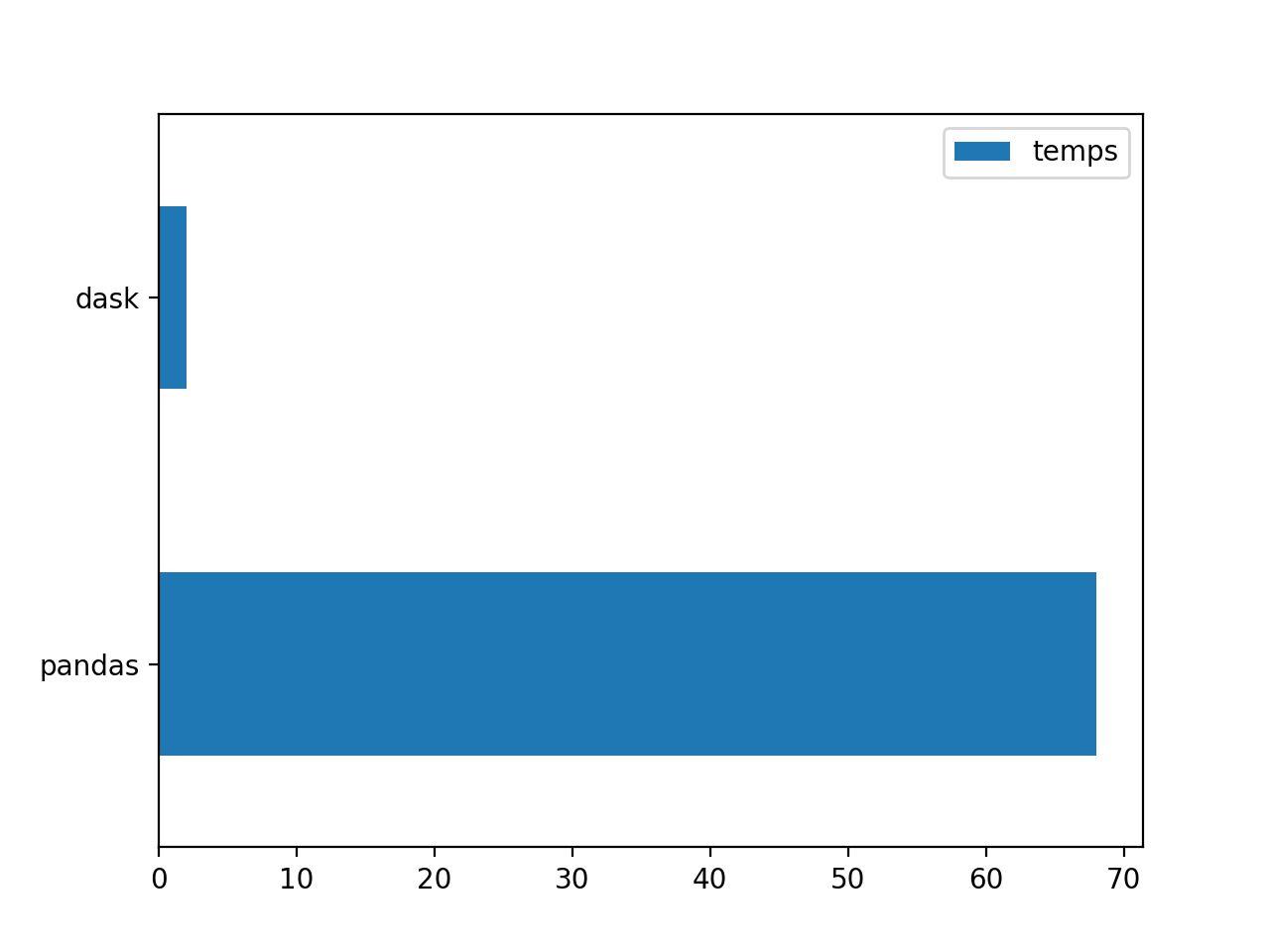
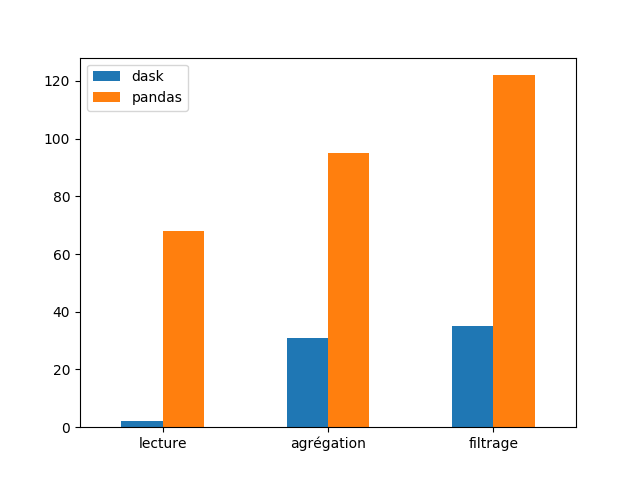
Les résultats montrent une grande différence de performance entre les deux librairies. Pandas a été trente fois plus lent que Dask dans la lecture et création du DataFrame. Dask a réussit à faire tout le traitement en 2 secondes contre 68 secondes pour Pandas.
Round 2 : Traitements des données
Après le premier test de lecture, avec des écarts de performance très importants, il semble judicieux de faire les mêmes comparaisons sur des opérations plus complexes et récurrentes. Bien que la lecture soit une étape obligatoire pour toute analyse de données, il est important de vérifier l’évolution des écarts de performance sur des opérations d’agrégation et de filtrage de données.
Agrégation :
En suivant la même démarche, j’ai écrit deux fonctions équivalentes afin de calculer la population totale des sites par pays.
import dask.dataframe as dd
import pandas as pd
COLUMNS = {0: 'geonames_id', 1: 'name', 2: 'asciiname', 3: 'alternatenames', 4: 'latitude', 5: 'longitude',
6: 'feature_class', 7: 'feature_code', 8: 'country_code', 9: 'cc2', 10: 'admin1_code',
11: 'admin2_code', 12: 'admin3_code', 13: 'admin4_code', 14: 'population', 15: 'elevation',
16: 'dem', 17: 'timezone', 18: 'modification_date'}
def test_geonames_dask_groupby():
df = dd.read_csv('allCountries.txt', sep='\t',
dtype={9: 'object', 10: 'object', 11: 'object',
12: 'object', 13: 'object'},
header=None)
df = df.rename(columns=COLUMNS)
return df.groupby('country_code').population.sum().compute()
def test_geonames_pandas_groupby():
df = pd.read_csv('allCountries.txt', sep='\t',
dtype={9: 'object', 10: 'object', 11: 'object',
12: 'object', 13: 'object'},
header=None)
df = df.rename(columns=(COLUMNS))
return df.groupby('country_code').population.sum()
Filtrage :
Pour le test de filtrage, j’ai écrit deux fonctions afin d’obtenir l’ensemble des sites situés à plus de deux mille mètres d’altitude.
import dask.dataframe as dd
import pandas as pd
COLUMNS = {0: 'geonames_id', 1: 'name', 2: 'asciiname', 3: 'alternatenames', 4: 'latitude', 5: 'longitude',
6: 'feature_class', 7: 'feature_code', 8: 'country_code', 9: 'cc2', 10: 'admin1_code',
11: 'admin2_code', 12: 'admin3_code', 13: 'admin4_code', 14: 'population', 15: 'elevation',
16: 'dem', 17: 'timezone', 18: 'modification_date'}
def test_geonames_dask_filtering():
df = dd.read_csv('allCountries.txt', sep='\t',
dtype={9: 'object', 10: 'object', 11: 'object',
12: 'object', 13: 'object'},
header=None)
df = df.rename(columns=COLUMNS)
df = df[~df.elevation.isna()].compute()
df = df[df.elevation > 2000]
return df[['name', 'longitude', 'latitude', 'country_code', 'elevation']].head()
def test_geonames_pandas_filtering():
df = pd.read_csv('allCountries.txt', sep='\t',
dtype={9: 'object', 10: 'object', 11: 'object',
12: 'object', 13: 'object'},
header=None)
df = df.rename(columns=COLUMNS)
df = df[~df.elevation.isna()]
df = df[df.elevation > 2000]
return df[['name', 'longitude', 'latitude', 'country_code', 'elevation']].head()
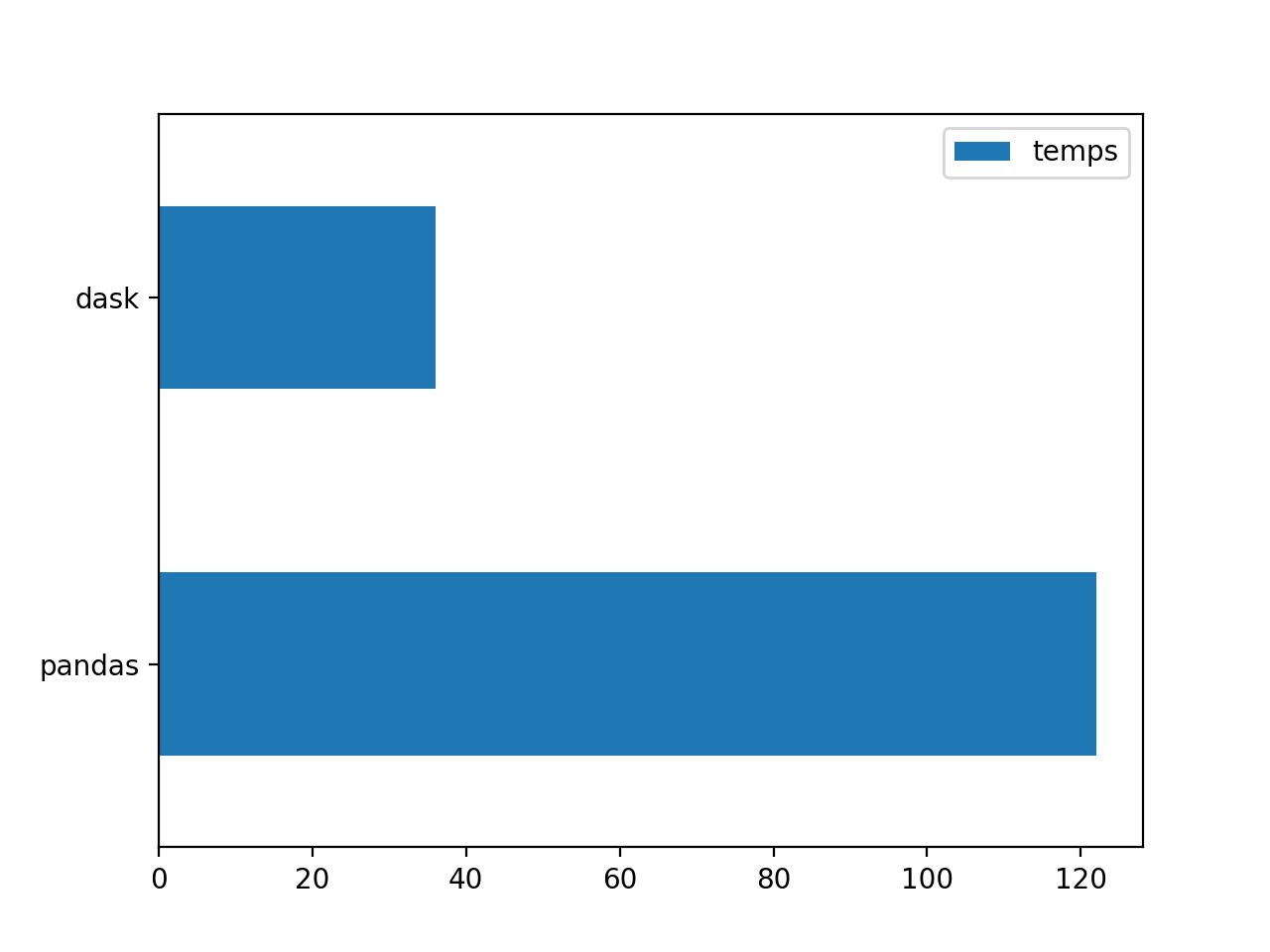
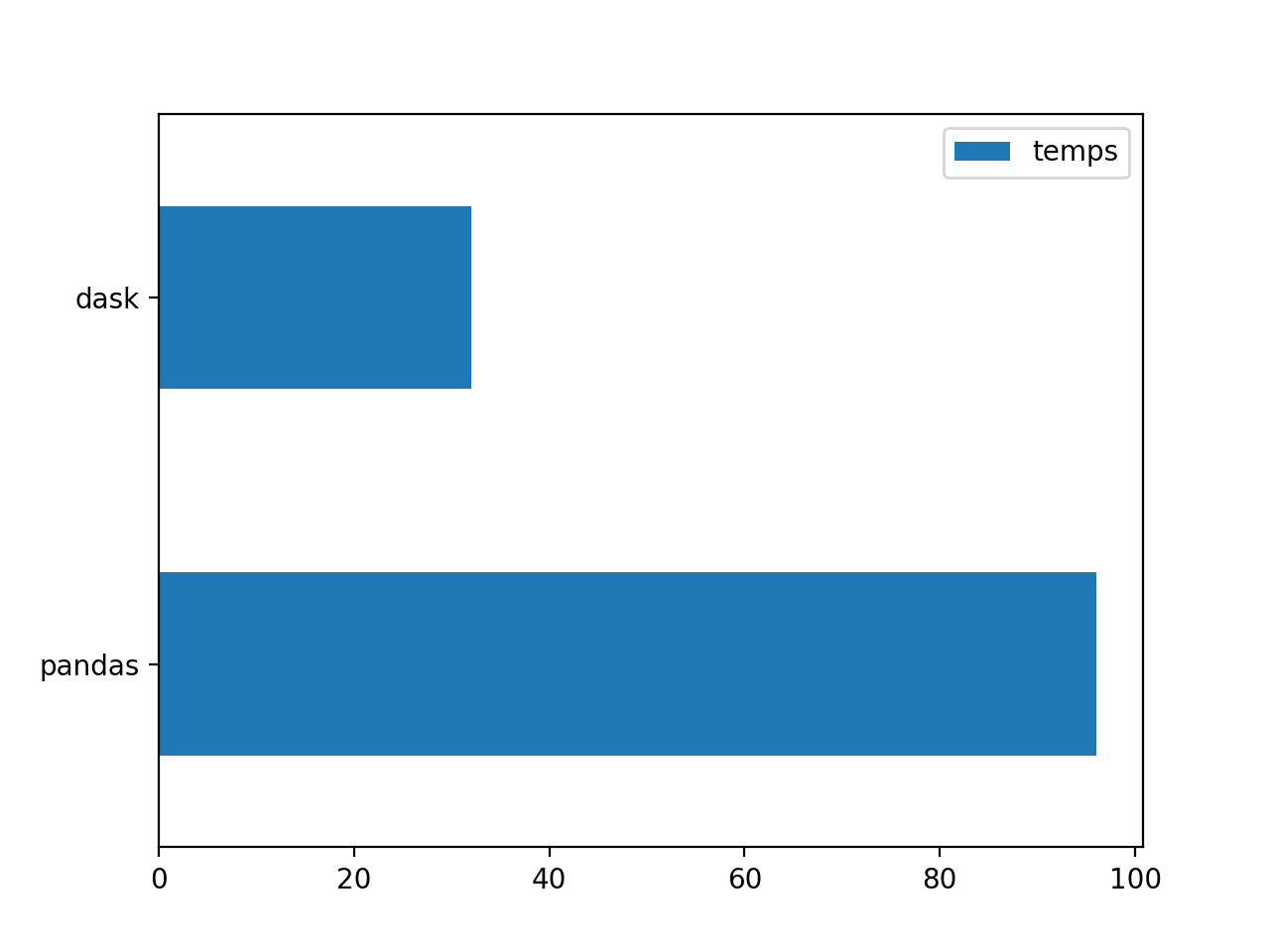
Les écarts de performance entre les librairies sont moins importants mais ils restent considérables. Sur le test d’agrégation, Dask finit les traitements en 32 secondes contre 96 pour Pandas. Concernant le filtrage, Dask finalise au bout de 36 secondes contre 122 pour Pandas. La performance de Dask est environ trois fois plus rapide que celle de Pandas.
Round 3 : lecture de fichier Excel
Dans cette dernière partie, J’ai voulu comparer la performance des libraries sur des fichiers Excel. L’objectif est de mettre en évidence les différence de performance en fonction du format de la source.
import dask.dataframe as dd
import pandas as pd
from dask.delayed import delayed
def test_dask_excel():
parts = delayed(pd.read_excel)('allCountries.xlsx', sheet_name='Sheet1')
return dd.from_delayed(parts)
def test_pandas_excel():
return pd.read_excel('allCountries.xlsx', sheet_name='Sheet1')
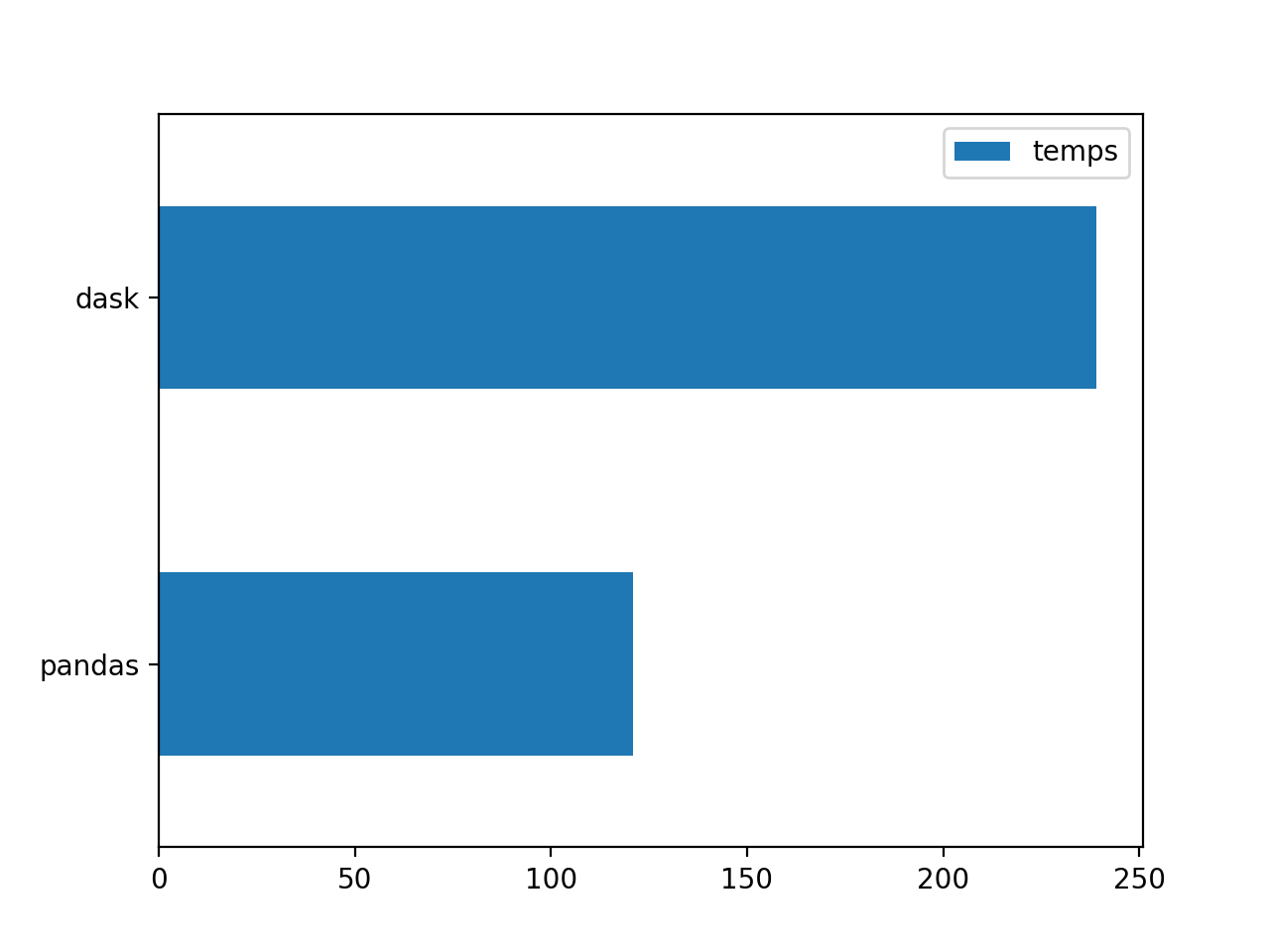
Dans ce dernier test on retrouve de résultats différents à ceux des deux permiers tests.
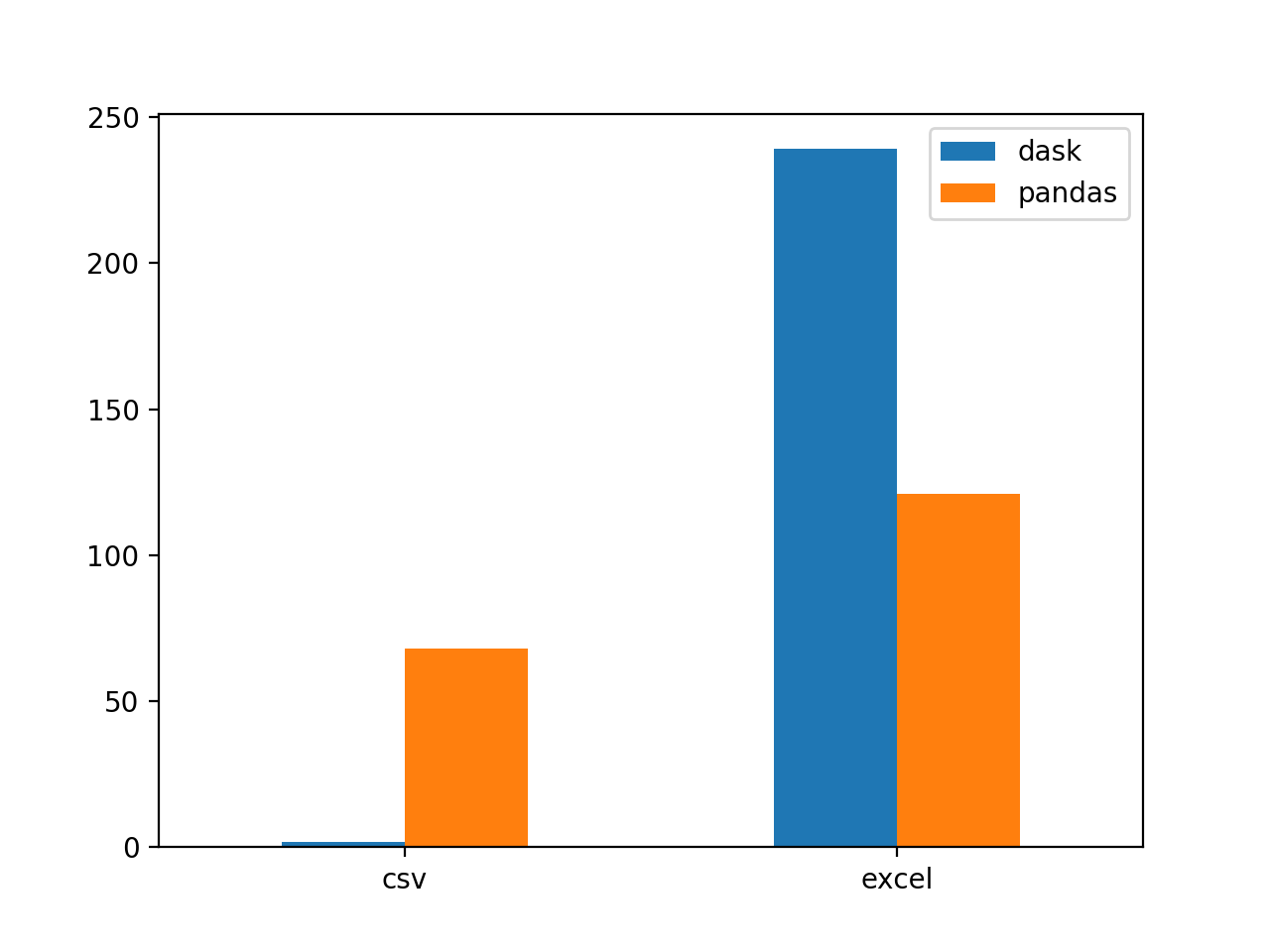
Pandas est environ deux fois rapide. Par ailleurs, Dask fait appel à une fonction Pandas pour la lecture d’un Excel
dask.delayed(pd.read_excel)('/path', sheet_name='name').
Concernant les différences de temps de calcul en fonction du format des fichiers source, on constate que le format txt/csv accelère considérablement le temps de traitement. On passe de 121 secondes à 68 secondes avec Pandas. Avec Dask l’écart est encore plus grand, on passe de 239 secondes à 2 scondes. Compte tenu des résultats, on pourait se poser la question suivante : doit-on travailler avec des fichiers Excel ?
Vainqueur : Dask, loser : Excel.
Les résultats de ces tests de comparaison permettent de faire les conclusions suivantes :
Dask est une librairie facilement adaptable aux projets développés avec Pandas. La syntaxe, la logique ainsi que les outputs, très similaires à ceux de Pandas, permettent de prendre facilement en main la librairie. Les temps de calcul avec les DataFrames Dask sont plus rapides. Des traitements récurrents sur les dataframes, comme les groupby et les filtres, montrent des résultats environ trois fois plus performants.
Les gains en performance sur des datasets de plus d’un gigaoctet sont très importants. Cependant, Pandas connaît de ralentissements avec des fichiers de taille inférieure à un gigaoctet. Il est sans doute judicieux de faire des tests sur des datasets de plusieurs centaines de megaoctet afin de définir un seuil adéquat pour le passage en Dask.
Finalement, il est important de répondre à la question concernant les fichiers Excel car les résultats sont très clairs. Il ne semble pas judicieux d’avoir des fichiers Excel comme source de données, indépendamment de la librairie utilisée. Ces derniers demandent plus d’espace de stockage, mais surtout, ils ralentissent les traitements des données. Un passage au format CSV aura un impact significatif dans les performances pour charger vos données.
Cette entrée a été publiée dans programmation avec comme mot(s)-clef(s) pandas, dask, python
Les articles suivant pourraient également vous intéresser :
- Principes SOLID et comment les appliquer en Python par Mariana ROLDAN VELEZ
- Pydantic, la révolution de python ? par Pablo Abril
- Comment utiliser les fixtures pytest en autouse tout en maîtrisant ses effets de bord ? par Amaury Boin
- Améliorer l'architecture d'une application Vue grâce à la composition API par Pierre Assemat
- Wrap like an Egyptian par Khaled FAYSAL
Vos commentaires
Très bon article Juan, simple et facile à lire, et surtout interessant, Bravo
Et Modin également qui a l’avantage d’avoir une API identique à celle de pandas !
Get faster pandas with Modin, even on your laptops.
Intéressant mais les comparaisons pour le traitement de données ne sont pas vraiment correctes. Il faudrait enlever le temps de lecture du temps total. Par example pour l’aggrégation, si on enlève le temps de lecture, le temps de traitement est presque identique (à vue d’oeil) entre les deux…
Parfait!!! Puis je savoir comment faites vous pour récupérer le fichier countries? Merci
La BD geonames peut être téléchargée ici : https://www.geonames.org/
Il faudrait préciser le type de machine sur laquelle tout cela tourne. De plus il me semble que pour Dask, le premier exemple est trompeur, je ne crois pas que Dask charge en mémoire s’il n’y a pas d’appel à compute.
Postez votre commentaire :
Votre commentaire a bien été envoyé ! Il sera affiché une fois que nous l'aurons validé.
Vous devez activer le javascript ou avoir un navigateur récent pour pouvoir commenter sur ce site.